Самые новые ссылки

|
|

|
|
Раздел: Генетика
|
 Как складывался генофонд славян и балтов
Как складывался генофонд славян и балтов 
|
Хитов: 448
 2016/2/14
2016/2/14
|
 : Олег Балановский, Надежда Маркина : Олег Балановский, Надежда Маркина
Реконструкция генетической и лингвистической истории балто-славянских популяций
Эта наиболее полная работа по генофонду славянских и балтских народов подводит итоги многолетних исследований. Для реконструкции давней истории народов, говорящих на родственных языках, применен междисциплинарный подход. Генетики и лингвисты проследили пути формирования генофонда всех групп славян и балтов одновременно по трем генетическим системам: по Y-хромосоме (отцовские линии наследования), по митохондриальной ДНК (материнские линии наследования) и по широгеномным данным об аутосомных маркерах (где отцовские и материнские линии представлены равноправно). Прослежено, какие местные популяции впитывал генофонд славян при их расселении по Европе: именно этот глубинный субстрат сформировал основные различия генофондов разных ветвей славян. Корреляция генетического разнообразия с лингвистическим оказалась велика, но еще больше — с географическим соседством популяций. Результатом исследования стало и уточнение древа балто-славянских языков.
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Исторические ресурсы в сети
|
 Балто-славянская языковая общность
Балто-славянская языковая общность 
|
Хитов: 693
 2015/10/9
2015/10/9
|
 : В семье индоевропейских языков особенно близки друг другу славянские и балтийские языки (как иранские и индоарийские, кельтские и италийские). : В семье индоевропейских языков особенно близки друг другу славянские и балтийские языки (как иранские и индоарийские, кельтские и италийские).
Близость балтийских и славянских языков проявляется в регулярных звуковых соответствиях, в сходстве форм словоизменения и словообразования, в общности большинства слов, обозначающих окружающий мир, людей, их отношения и деятельность в условиях общинно-родового строя.
При этом восстанавливаемое для славянских языков исторически исходное праславянское оформление слов, как правило, совпадает с их оформлением в исторически засвидетельствованных балтийских языках. Так, например, восстанавливая для славянского слова "верх" (древнерусское вьрхъ) праформу *virs-us, мы находим ее в латышском virs-us и литовском virs-us. Восстанавливая для славянского "сын" (древнерусское сынъ) праформу *sun-us, мы находим ее в литовском sun-us и т. д. [Восстанавливая для славянского "цена" (родственное, вероятно, глаголу "каять" и означающее, видимо, плату за преступление) праформу *kai-n-a, мы находим ее в латышском kaina. В очень большом числе случаев, таким образом, славянские слова и формы выглядят как преобразованные балтийские.
Эти уникальные внутри индоевропейской семьи отношения между языками, при надлежащими к разным группам, до сих пор не получили общепринятого исторического объяснения.
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Генетика
|
 Uniparental Genetic Heritage of Belarusians: Encounter of Ra
Uniparental Genetic Heritage of Belarusians: Encounter of Ra 
|
Хитов: 5698
 2015/10/9
2015/10/9
|
 : Alena Kushniarevich, Larysa Sivitskaya, Nina Danilenko, Tadeush Novogrodskii, Iosif Tsybovsky, Anna Kiseleva, Svetlana Kotova, Gyaneshwer Chaubey, Ene Metspalu, Hovhannes Sahakyan, Ardeshir Bahmanimehr, Maere Reidla, Siiri Rootsi, Jüri Parik, Tuuli Reisberg, Alessandro Achilli, Baharak Hooshiar Kashani, Francesca Gandini, Anna Olivieri, Doron M. Behar, Antonio Torroni, Oleg Davydenko, Richard Villems : Alena Kushniarevich, Larysa Sivitskaya, Nina Danilenko, Tadeush Novogrodskii, Iosif Tsybovsky, Anna Kiseleva, Svetlana Kotova, Gyaneshwer Chaubey, Ene Metspalu, Hovhannes Sahakyan, Ardeshir Bahmanimehr, Maere Reidla, Siiri Rootsi, Jüri Parik, Tuuli Reisberg, Alessandro Achilli, Baharak Hooshiar Kashani, Francesca Gandini, Anna Olivieri, Doron M. Behar, Antonio Torroni, Oleg Davydenko, Richard Villems
PLOS
Published: June 13, 2013
DOI: 10.1371/journal.pone.0066499
Uniparental Genetic Heritage of Belarusians: Encounter of Rare Middle Eastern Matrilineages with a Central European Mitochondrial DNA Pool
N1c(Tat) along with its much less frequent sister group N1b(P43) (previously N2), detected in Belarusians indicate an ancient patrilineal gene flow from the north Eurasia westward, yet in the context of studied here populations is best explained by partially shared Y-chromosomal ancestry of Belarusians and their northern neighbors, Lithuanians and Latvians, among whom N1c(Tat) reaches frequencies above 40% [20], [21], [26], [58].
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Генетика
|
 Почему у славян и прибалтов так много общего
Почему у славян и прибалтов так много общего 
|
Хитов: 1472
 2015/10/9
2015/10/9
|
 : Елена Кушнеревич (Эстонский биоцентр, Тарту) так прокомментировала результаты: : Елена Кушнеревич (Эстонский биоцентр, Тарту) так прокомментировала результаты:
«Судя по всему, генетический ландшафт Европы и, в частности, Восточной Европы сформировался задолго до великого переселения народов, поэтому сравнительно недавние миграции, пусть даже и массовые, уже не могли серьезно перестроить региональные генетические структуры: плотность населения была уже слишком высокой, и чтобы оставить свой хорошо различимый след, мигрантам нужно было бы уничтожить слишком много автохтонного населения».
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Генетика
|
 Genetic Discontinuity Between Local Hunter-Gatherers and Cen
Genetic Discontinuity Between Local Hunter-Gatherers and Cen 
|
Хитов: 9718
 2015/10/9
2015/10/9
|
 : Genetic Discontinuity Between Local Hunter-Gatherers and Central Europe's First Farmers : Genetic Discontinuity Between Local Hunter-Gatherers and Central Europe's First Farmers
Barbara Bramanti Mark G Thomas W Haak Martina Unterländer Pia Carolin Jores Kristiina Tambets I Antanaitis-Jacobs Miriam Noël Haidle Rimantas Jankauskas C-J Kind Friedrich Lüth Thomas Terberger J Hiller S Matsumura P Forster J Burger
Institute for Anthropology, University of Mainz, Mainz, Germany.
Science (Impact Factor: 33.61). 10/2009; 326(5949):137-40. DOI: 10.1126/science.1176869
Source: PubMed
ABSTRACT
After the domestication of animals and crops in the Near East some 11,000 years ago, farming had reached much of central Europe by 7500 years before the present. The extent to which these early European farmers were immigrants or descendants of resident hunter-gatherers who had adopted farming has been widely debated. We compared new mitochondrial DNA (mtDNA) sequences from late European hunter-gatherer skeletons with those from early farmers and from modern Europeans. We find large genetic differences between all three groups that cannot be explained by population continuity alone. Most (82%) of the ancient hunter-gatherers share mtDNA types that are relatively rare in central Europeans today. Together, these analyses provide persuasive evidence that the first farmers were not the descendants of local hunter-gatherers but immigrated into central Europe at the onset of the Neolithic.
Genetic Discontinuity Between Local Hunter-Gatherers and Central Europe's First Farmers (PDF Download Available). Available from: http://www.researchgate.net/publication/26788823_Genetic_Discontinuity_Between_Local_Hunter-Gatherers_and_Central_Europe's_First_Farmers [accessed Oct 9, 2015].
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Генетика
|
 Как складывался генофонд славян и балтов
Как складывался генофонд славян и балтов 
|
Хитов: 2695
 2015/10/9
2015/10/9
|
 : Олег Балановский, Надежда Маркина : Олег Балановский, Надежда Маркина
Реконструкция генетической и лингвистической истории балто-славянских популяций
Эта наиболее полная работа по генофонду славянских и балтских народов подводит итоги многолетних исследований. Для реконструкции давней истории народов, говорящих на родственных языках, применен междисциплинарный подход. Генетики и лингвисты проследили пути формирования генофонда всех групп славян и балтов одновременно по трем генетическим системам: по Y-хромосоме (отцовские линии наследования), по митохондриальной ДНК (материнские линии наследования) и по широгеномным данным об аутосомных маркерах (где отцовские и материнские линии представлены равноправно). Прослежено, какие местные популяции впитывал генофонд славян при их расселении по Европе: именно этот глубинный субстрат сформировал основные различия генофондов разных ветвей славян. Корреляция генетического разнообразия с лингвистическим оказалась велика, но еще больше — с географическим соседством популяций. Результатом исследования стало и уточнение древа балто-славянских языков.
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Генетика
|
 Genetic Heritage of the Balto-Slavic Speaking Populations: A
Genetic Heritage of the Balto-Slavic Speaking Populations: A 
|
Хитов: 8813
 2015/10/9
2015/10/9
|
 : Alena Kushniarevich, Olga Utevska, Marina Chuhryaeva, Anastasia Agdzhoyan, Khadizhat Dibirova, Ingrida Uktveryte, Märt Möls, Lejla Mulahasanovic, Andrey Pshenichnov, Svetlana Frolova, Andrey Shanko, Ene Metspalu, Maere Reidla, Kristiina Tambets, Erika Tamm, Sergey Koshel, Valery Zaporozhchenko, Lubov Atramentova, Vaidutis Kučinskas, Oleg Davydenko, Olga Goncharova, Irina Evseeva, Michail Churnosov, Elvira Pocheshchova, Bayazit Yunusbayev, Elza Khusnutdinova, Damir Marjanović, Pavao Rudan, Siiri Rootsi, Nick Yankovsky, Phillip Endicott, Alexei Kassian, Anna Dybo, The Genographic Consortium, Chris Tyler-Smith, Elena Balanovska, Mait Metspalu, Toomas Kivisild, Richard Villems, Oleg Balanovsky : Alena Kushniarevich, Olga Utevska, Marina Chuhryaeva, Anastasia Agdzhoyan, Khadizhat Dibirova, Ingrida Uktveryte, Märt Möls, Lejla Mulahasanovic, Andrey Pshenichnov, Svetlana Frolova, Andrey Shanko, Ene Metspalu, Maere Reidla, Kristiina Tambets, Erika Tamm, Sergey Koshel, Valery Zaporozhchenko, Lubov Atramentova, Vaidutis Kučinskas, Oleg Davydenko, Olga Goncharova, Irina Evseeva, Michail Churnosov, Elvira Pocheshchova, Bayazit Yunusbayev, Elza Khusnutdinova, Damir Marjanović, Pavao Rudan, Siiri Rootsi, Nick Yankovsky, Phillip Endicott, Alexei Kassian, Anna Dybo, The Genographic Consortium, Chris Tyler-Smith, Elena Balanovska, Mait Metspalu, Toomas Kivisild, Richard Villems, Oleg Balanovsky
PLOS
Published: September 2, 2015
DOI: 10.1371/journal.pone.0135820
Genetic Heritage of the Balto-Slavic Speaking Populations: A Synthesis of Autosomal, Mitochondrial and Y-Chromosomal Data
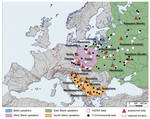
The Slavic branch of the Balto-Slavic sub-family of Indo-European languages underwent rapid divergence as a result of the spatial expansion of its speakers from Central-East Europe, in early medieval times. This expansion–mainly to East Europe and the northern Balkans–resulted in the incorporation of genetic components from numerous autochthonous populations into the Slavic gene pools. Here, we characterize genetic variation in all extant ethnic groups speaking Balto-Slavic languages by analyzing mitochondrial DNA (n = 6,876), Y-chromosomes (n = 6,079) and genome-wide SNP profiles (n = 296), within the context of other European populations. We also reassess the phylogeny of Slavic languages within the Balto-Slavic branch of Indo-European. We find that genetic distances among Balto-Slavic populations, based on autosomal and Y-chromosomal loci, show a high correlation (0.9) both with each other and with geography, but a slightly lower correlation (0.7) with mitochondrial DNA and linguistic affiliation. The data suggest that genetic diversity of the present-day Slavs was predominantly shaped in situ, and we detect two different substrata: ‘central-east European’ for West and East Slavs, and ‘south-east European’ for South Slavs. A pattern of distribution of segments identical by descent between groups of East-West and South Slavs suggests shared ancestry or a modest gene flow between those two groups, which might derive from the historic spread of Slavic people.
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|
|
|
Раздел: Генетика
|
 Как складывался генофонд славян
Как складывался генофонд славян 
|
Хитов: 632
 2015/10/9
2015/10/9
|
 : Опубликована наиболее полная на сегодняшний день работа по изучению генофонда славян и балтов, в которой использован синтез генетики и лингвистики. Генетики и лингвисты изучили балто-славянские народы в сравнении с другими народами Европы. Им удалось проследить, как формировался генофонд славян - при распространении по Европе славяне смешивались с местными популяциями, которые составили глубинный субстрат генофондов, отличающий разные ветви славян друг от друга. Исследователи обнаружили связь генетического разнообразия с лингвистическим и еще более сильную связь - с географическим расположением популяций. : Опубликована наиболее полная на сегодняшний день работа по изучению генофонда славян и балтов, в которой использован синтез генетики и лингвистики. Генетики и лингвисты изучили балто-славянские народы в сравнении с другими народами Европы. Им удалось проследить, как формировался генофонд славян - при распространении по Европе славяне смешивались с местными популяциями, которые составили глубинный субстрат генофондов, отличающий разные ветви славян друг от друга. Исследователи обнаружили связь генетического разнообразия с лингвистическим и еще более сильную связь - с географическим расположением популяций.
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|

|
|
Раздел: Генетика
|
 Y chromosome and mitochondrial DNA variation in Lithuanians.
Y chromosome and mitochondrial DNA variation in Lithuanians. 
|
Хитов: 2131
 2015/8/2
2015/8/2
|
 : Kasperaviciūte D., Kucinskas V., Stoneking M. Y chromosome and mitochondrial DNA variation in Lithuanians. : Kasperaviciūte D., Kucinskas V., Stoneking M. Y chromosome and mitochondrial DNA variation in Lithuanians.
Author information
Department of Human and Medical Genetics, Faculty of Medicine, Vilnius University, Santariskiu str. 2, LT-08661 Vilnius, Lithuania. dalia.kasperaviciute@gf.vu.lt
Abstract
The genetic composition of the Lithuanian population was investigated by analysing mitochondrial DNA hypervariable region 1, RFLP polymorphisms and Y chromosomal biallelic and STR markers in six ethnolinguistic groups of Lithuanians, to address questions about the origin and genetic structure of the present day population. There were no significant genetic differences among ethnolinguistic groups, and an analysis of molecular variance confirmed the homogeneity of the Lithuanian population. MtDNA diversity revealed that Lithuanians are close to both Slavic (Indo-European) and Finno-Ugric speaking populations of Northern and Eastern Europe. Y-chromosome SNP haplogroup analysis showed Lithuanians to be closest to Latvians and Estonians. Significant differences between Lithuanian and Estonian Y chromosome STR haplotypes suggested that these populations have had different demographic histories. We suggest that the observed pattern of Y chromosome diversity in Lithuanians may be explained by a population bottleneck associated with Indo-European contact. Different Y chromosome STR distributions in Lithuanians and Estonians might be explained by different origins or, alternatively, be the result of some period of isolation and genetic drift after the population split.
|
| Голосовать | Сообщить о потерянной ссылке | Написать друзьям |
|
|
|
|


